Productes naturals i SARS-CoV-2

La lluita contra la pandèmia de COVID-19 és un desafiament científic a escala internacional per trobar estratègies per combatre la infecció viral i prevenir-ne les conseqüències. A més de les vacunes i els medicaments aprovats per les autoritats sanitàries, és vital trobar nous tractaments per lluitar contra la infecció vírica un cop s’ha produït.
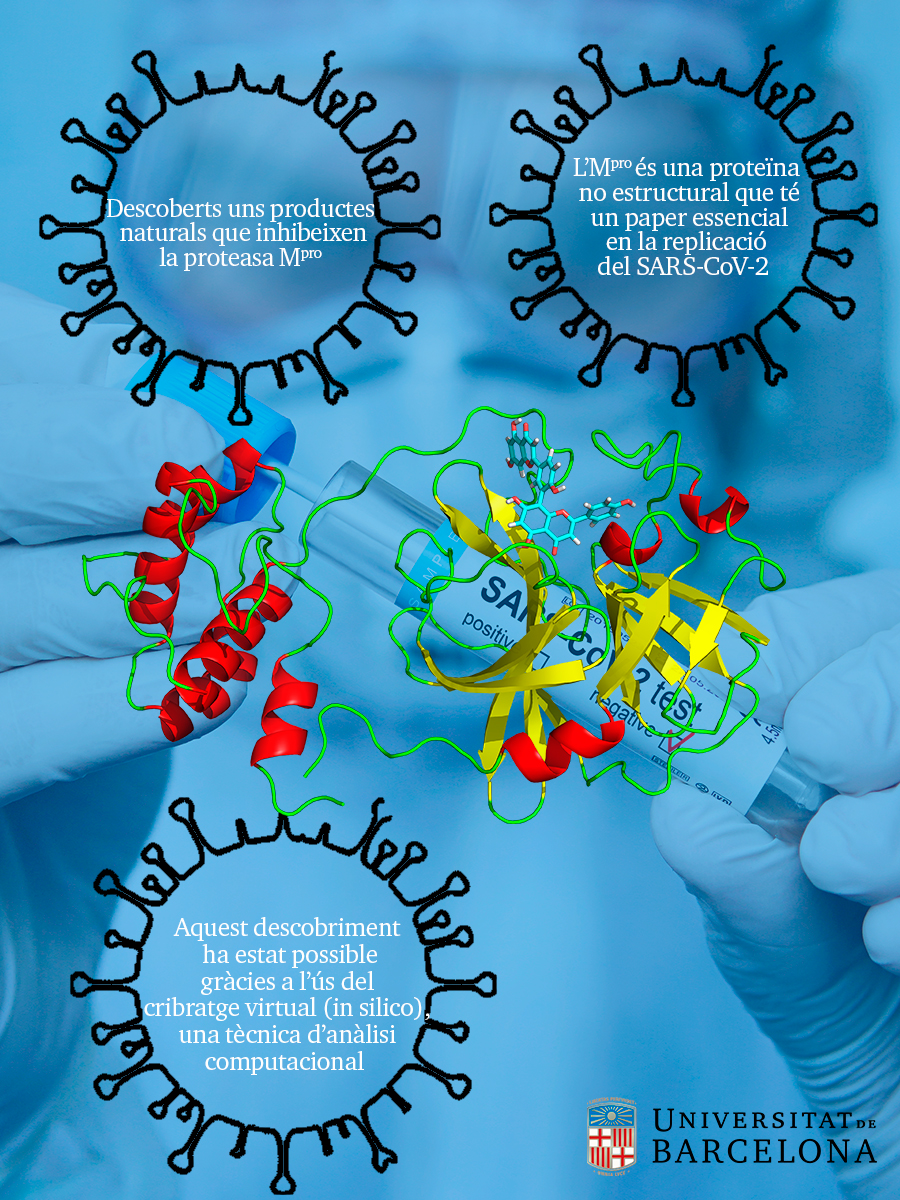
Un estudi publicat a la revista Journal of Chemical Information and Modeling ha identificat una sèrie de productes naturals que són capaços d’inhibir l’Mpro, la proteasa principal del virus SARS-CoV-2. L’Mpro és una proteïna no estructural que té un paper essencial en la replicació i la transcripció d’aquest virus, i es considera una potencial diana terapèutica ja que si es pogués inhibir podria evitar la progressió del virus.
El treball està dirigit pel catedràtic Jaime Rubio Martínez, de la Facultat de Química i de l’Institut de Química Teòrica i Computacional (IQTC) de la Universitat de Barcelona, i pot obrir perspectives per dissenyar noves estratègies terapèutiques en la lluita contra la COVID-19. Aquest nou avenç científic es basa en l’ús del cribratge virtual (in silico), una tècnica d’anàlisi computacional de molècules emmagatzemades en bases de dades per identificar i seleccionar compostos candidats de potencial interès farmacològic.
Cribratge virtual: a la recerca de futurs fàrmacs
Avui dia, l’abordament clínic de la COVID-19 consisteix en el tractament simptomàtic mitjançant agents antiinflamatoris (per exemple, la dexametasona o inhibidors de citocines) combinats amb antibiòtics per tractar infeccions secundàries. Més recentment també s’ha aprovat l’antiviral ritonavir, però encara hi ha necessitat de nous medicaments. En el context de la salut global, és urgent desenvolupar teràpies antivirals específiques contra el SARS-CoV-2 i això ha portat a posar en marxa diverses estratègies per identificar compostos bioactius que es puguin utilitzar com a agents terapèutics (inclosos els fàrmacs disponibles i els productes naturals).
En aquest context, el cribratge virtual és un procediment fiable, ràpid i eficient per descobrir compostos bioactius de grans col·leccions químiques contra un objectiu molecular específic o diana.
En el marc d’aquest treball, l’equip ha dut a terme un cribratge virtual de la base de dades Selleck de productes naturals —una biblioteca química amb prop de 2.000 compostos— per a un conjunt de diverses configuracions de la proteasa Mpro del virus SARS-CoV-2, caracteritzades a partir d’un estudi de dinàmica molecular.
Com a resultat, el treball presenta en primer lloc la caracterització del perfil dinàmic de la proteasa Mpro en la forma apo per mitjà de simulacions convencionals (cMD) i de dinàmica molecular accelerada gaussiana (GaMD), i defineix una sèrie d’estructures representatives. Aquestes estructures es van utilitzar posteriorment per dur a terme l’acoblament molecular, un mètode bioinformàtic que permet predir i calcular amb tècniques computacionals la posició més favorable d’interacció entre un lligand i la seva proteïna diana. A continuació, es va desenvolupar un procés iteratiu amb què es va incrementar la longitud de les cMD dels complexos proteïna-lligand i es va calcular l’energia lliure d’unió per seleccionar els candidats més prometedors.
Tenint en compte els resultats, es van seleccionar onze compostos i es van provar in vitro per la seva capacitat d’inhibir la proteasa Mpro. A l’últim, del conjunt final es van identificar cinc candidats antivirals SARS-CoV-2, inhibidors de la proteasa Mpro, a partir de la base de dades de productes naturals.
Article de referència:
Rubio-Martínez, J. et al. «Discovery of diverse natural products as inhibitors of SARS-CoV-2 Mpro protease through virtual screening». Journal of Chemical Information and Modeling, novembre de 2021. DOI: 10.1021/acs.jcim.1c00951